中国学者破译产卵哺乳动物基因组
发布时间:2024年03月20日 作者:张艺 鲁明月
【摘要】产卵哺乳动物(单孔目)是兽类(有袋动物和真兽类动物)唯一现存的哺乳动物外类群,为哺乳动物进化提供了关键见解。在这里,我们生成并分析了鸭嘴兽(Ornithorhynchus anatinus)和短吻针鼹(Tachyglosus acuretus)的参考基因组,这两个物种代表了现存仅有的两个单孔目谱系。几乎完整的鸭嘴兽基因组组装将几乎整个基因组锚定在染色体上,显著提高了基因组的连续性和基因注解。结合我们的echidna序列,这两个物种的基因组,使我们能够检测到决定单孔目和哺乳动物进化的祖先和谱系特异性基因组变化。我们提供的证据表明,单孔目性染色体复合体起源于祖先的染色体环结构。人类常染色体同源物共有的多X染色体和多Y染色体之间异常广泛的相互作用可能促进了这种独特染色体复合体的形成。进一步的比较基因组分析揭示了单孔目和鲸目动物在触珠蛋白基因、泌乳基因和嗅觉和味觉化学感受受体基因方面的显著差异,这些基因是单孔目生态适应的基础。
【图表】
1、Fig1:单孔目的染色体组装与哺乳动物基因组进化
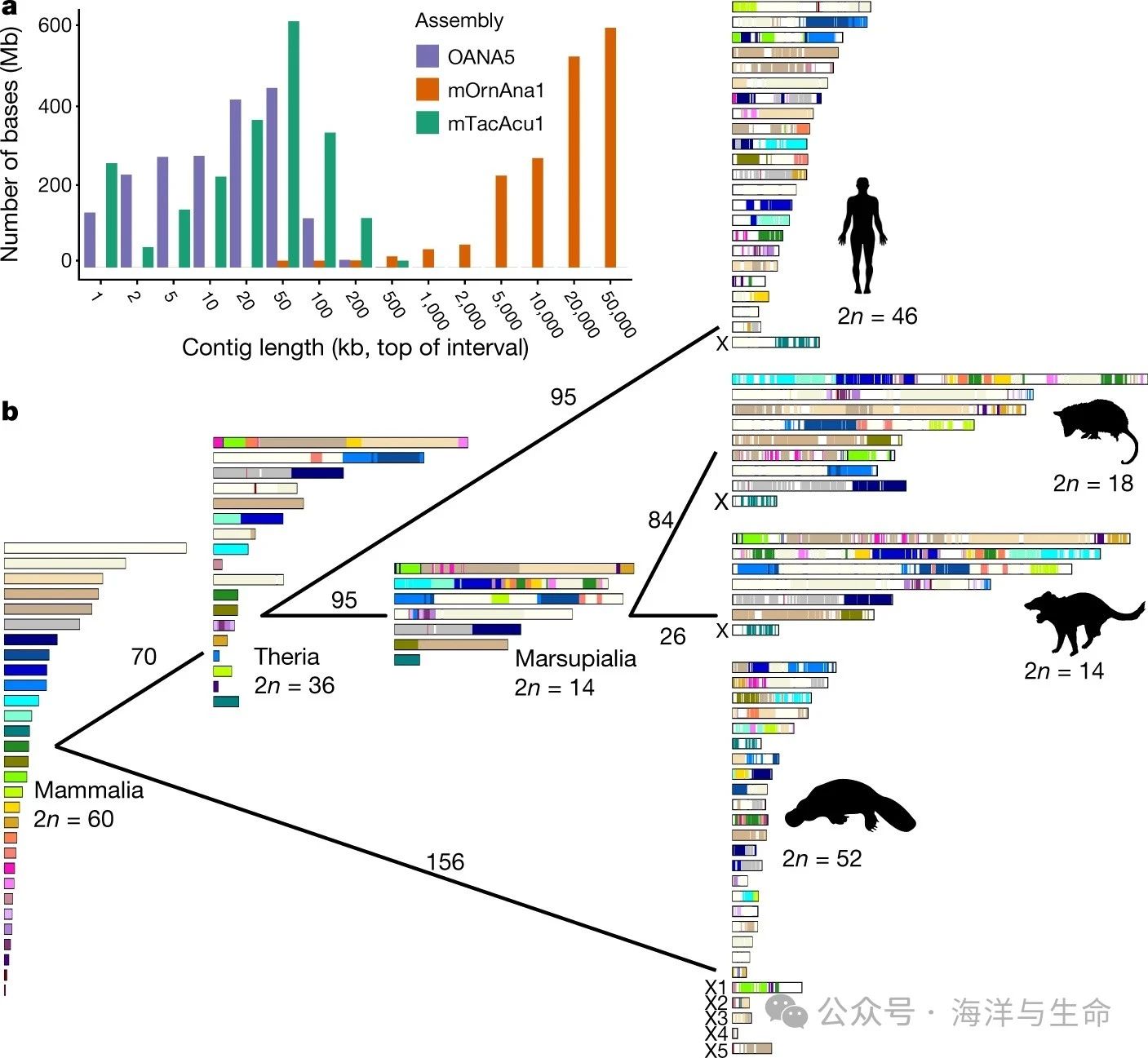
a,三个单孔目组合体之间的重叠群长度分布显示出鸭嘴兽组合体的序列连续性有了很大的改善,并且至少与针鼹组合体的质量相当。
b,哺乳动物核型进化轨迹。2n=60个祖先核型推断为哺乳动物的共同祖先。根据哺乳动物祖先的染色体来源对保存的块进行颜色编码。显示了每个分支的估计重排次数。
2、Fig2:鸭嘴兽性染色体的起源和进化
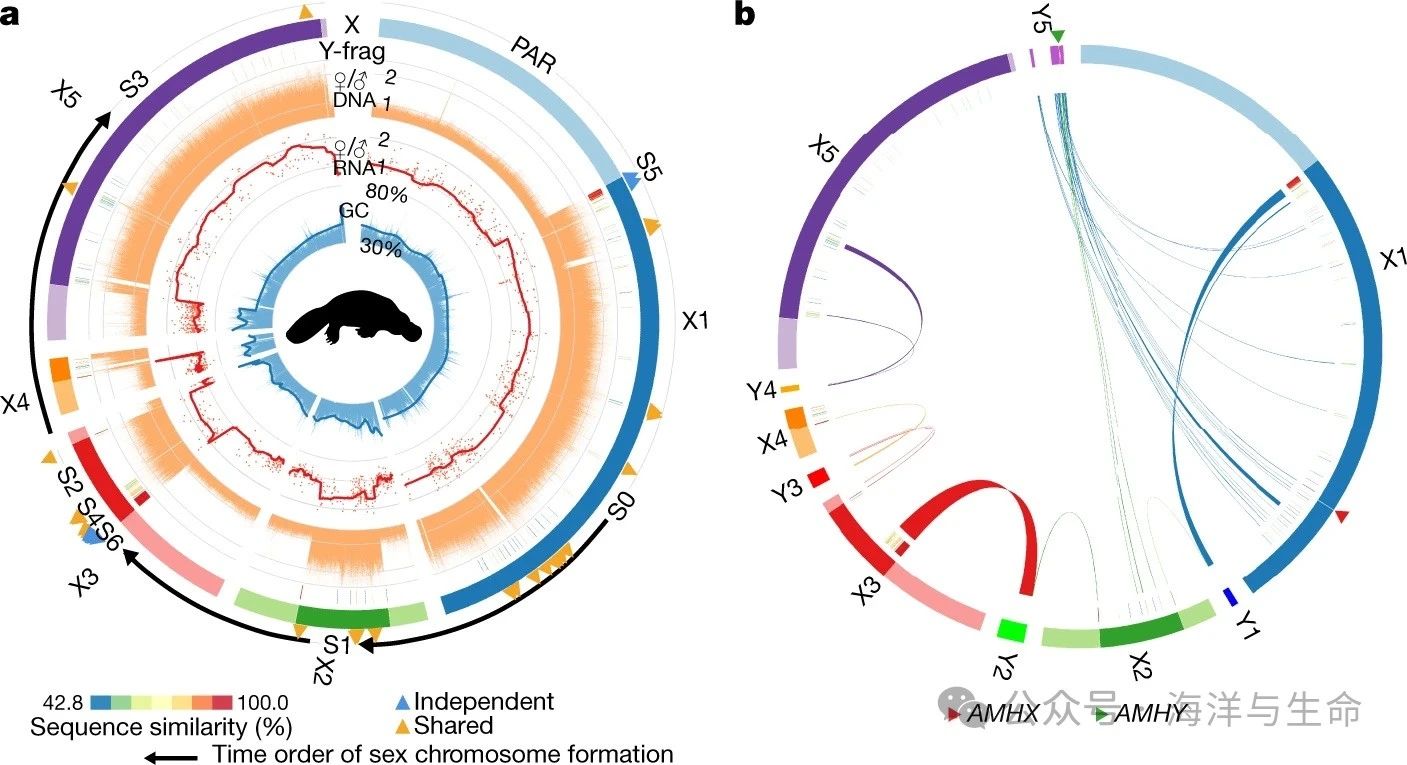
a,鸭嘴兽性染色体的基因组组成。从外圈到内圈:标记有PAR(浅色)和SDR(深色)的X染色体;SDR内组装的Y染色体片段显示出与同源X染色体的颜色标度序列差异水平;非重叠5kb窗口中短测序读数覆盖的女性与男性(F/M)比率;成年肾脏的F/M表达比率(每个红点是一个基因)和平滑的表达趋势;以及非重叠2kb窗口中的GC含量。此外,我们标记了在单孔目分化之前(“红色”,橙色三角形)或分化之后(“依赖性”,蓝色三角形)抑制重组的配子对在X染色体环上的位置。
b,鸭嘴兽X和Y染色体的同源性。特别地,Y5的大部分显示出与X1和X2的同源性,这表明鸭嘴兽性染色体的祖先环构象。我们还标记了假定的性别决定基因AMH的位置。
3、Fig3:鸭嘴兽性染色体之间的相互作用
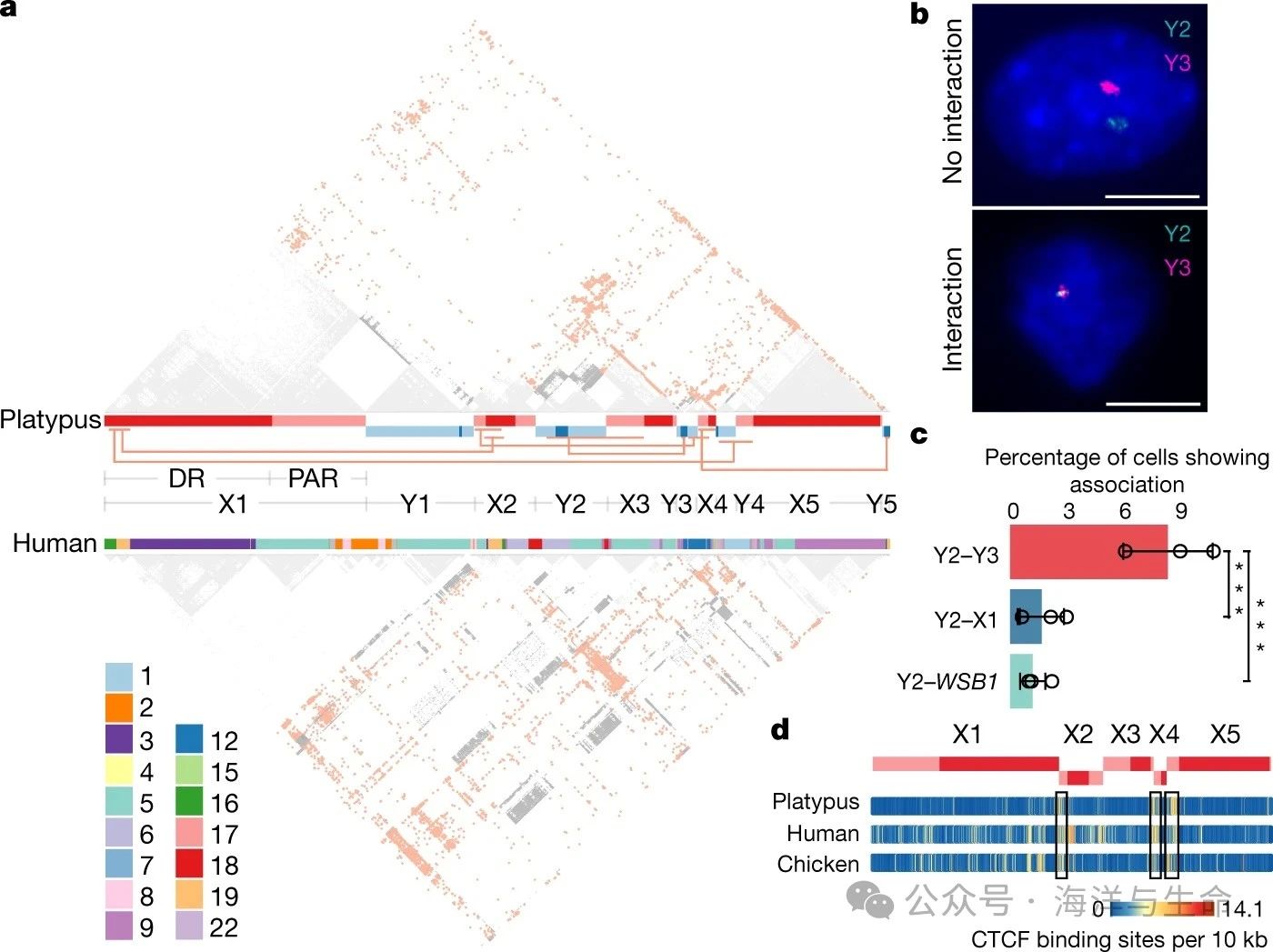
a,通过鸭嘴兽(上)和人类(下)肝组织的Hi-C数据检测到鸭嘴兽性染色体之间的染色体间相互作用。Hi-C面板之间的条形图显示了鸭嘴兽的性染色体及其在人类基因组中的直系同源物。灰色,染色体内相互作用;红色,染色体间相互作用。红线将具有显著高染色体间相互作用的区域连接起来。人类和鸭嘴兽性染色体的同源染色体片段及其Hi-C接触模式表明,哺乳动物的染色体间相互作用似乎是保守的。
b,用BAC探针FISH检测相间鸭嘴兽成纤维细胞中的性染色体Y2、Y3或X1以及常染色体17(WSB1)。实例显示染色体Y2和Y3之间没有相互作用(顶部,n=593,3个独立实验)和相互作用(底部,n=56,3个单独实验)。比例尺,10μm。
c,Y2和Y3之间的相互作用频率明显高于Y2和X1之间以及Y2和WSB1之间(17号染色体)。Y2–Y3三个独立复制实验的n=185206258个细胞,Y2–X1三个独立重复实验的n=258250205个细胞,以及Y2–WSB1三个独立实验的n=298262220个细胞。数据为平均值±s.d.***P<0.001(Y2–Y3与Y2–X1,P=0.0004675;Y2–Y1与Y2-WSB1,P=6.376×10−5),单侧Fisher精确检验。
d,推测的CTCF结合位点密度图显示其在鸭嘴兽、人类和鸡基因组的同源区域中富集。
4、Fig4:和单孔目生物特征相关的基因组特征
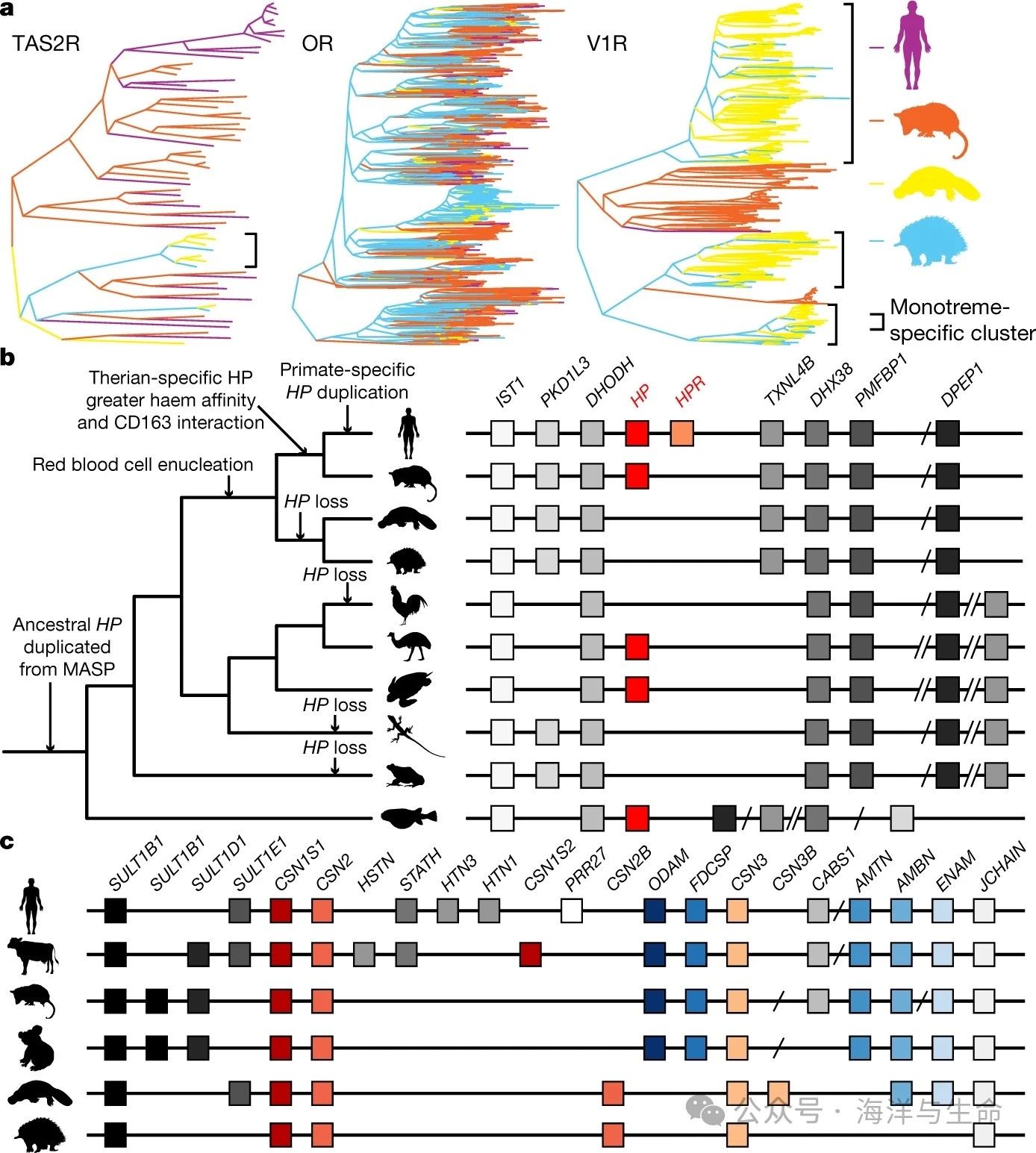
a,鸭嘴兽和针鼹TAS2R、OR和V1R基因数量的差异。
b,HP基因的系统发育和同源性。区域不是按比例绘制的。
c,酪蛋白周围区域(CSN基因)和祖先牙齿基因(ODAM、FDCSP、AMTN、AMBN和ENAM)的协同保护。
5、扩展数据图1:鸭嘴兽基因组组装和评估
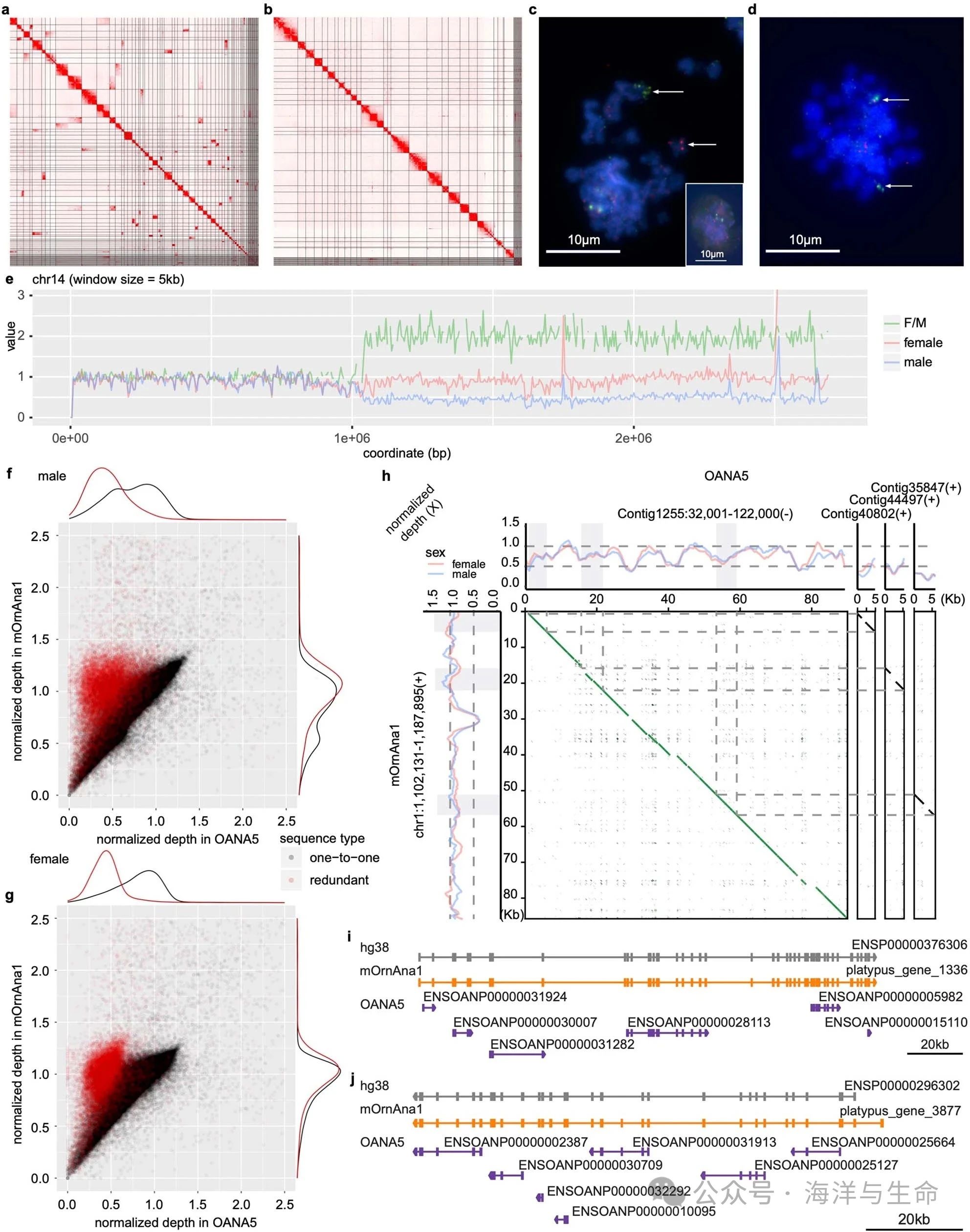
a,b,在(a)手动组装管理之前和之后的mOrnAna1的Hi-C二维juicebox图。支架之间的非对角线匹配表示潜在的漏接,而支架边界内的“空”区域表示漏接。gEVAL支持的手动组装策略显著改善了染色体规模支架内>96%的组装序列的排列。
c,d,在OANA5中,Super_Scaffold_40被错误地分配到第15号染色体,但从鸭嘴兽成纤维细胞中期传播的FISH将其映射到第13号染色体。c、 第15号染色体的BAC(绿色,顶部箭头)和Super_Scaffold_40探针(红色,底部箭头)的共杂交显示没有共定位(14个细胞核得分,2个独立实验)。插入,相间示例。d、 13号染色体的BAC(绿色)和Super_Scaffold_40探针(红色)的共杂交显示共定位(箭头,40个细胞核得分,5个独立实验)。比例尺,10μm。
e,OANA5支架染色体错位的一个例子。在5kb非重叠窗口中沿着OANA5 14号染色体的雌性与雄性(F/M)深度比、归一化雌性深度和归一化雄性深度。深度比、标准化女性深度和标准化男性深度均表明OANA5 14号染色体应为X染色体而非常染色体序列。
f,g,雄性(f)和雌性(g)中冗余序列和一对一序列的归一化深度分布。OANA5中的冗余序列(红色)可能是由于OANA5已测序个体的杂合物引起的组装伪影,因此在雄性和雌性中,OANA5的归一化深度为0.5倍,而mOrnAna1的归一化深度均为1倍。OANA5(黑色)中的一对一序列在OANA5和mOrnAna1中都具有1×标准化深度,这是按预期从两性映射的读数。每个点通过Mashmap表示OANA5和mOrnAna1之间的一个映射区域,并且每个点的归一化深度值被计算为OANA5中的映射区域和mOrnAna1中的平均深度。雄性中一对一序列密度图中的小峰指示候选X连锁序列。
h,OANA5中可能被解释为假重复的示例冗余序列Contig40802、Contig44497和Contig35847。通过FlexiDot在mOrnAna1染色体1的靶区与OANA5 Contig1255、Contig40802、Contig44497和Contig35847之间产生点图。候选冗余序列是映射到mOrnAna1染色体1中相同区域的序列,在点图中用虚线突出显示,在归一化深度图中用灰色突出显示。在500bp窗口中计算沿着每个序列的标准化雄性和雌性读取深度,并沿着每个序列绘制。尽管在mOrnAna1染色体1的区域中标准化深度总是在1左右,但在Contig40802、Contig44497、Contig35847和Contig1255中的对齐区域中,标准化深度下降了一半,这表明Contig40802Contig44497Contig35847Contig可能是OANA5中的冗余序列。
i,j,OANA5:CIT(i)和PBRM1(j)中的基因注释伪影的例子已经在OANA5(紫色)中被片段化为多个小的人工基因,但现在已经在mOrnAna1(橙色)中完全恢复。同源人类基因(灰色)也表明mOrnAna1而不是OANA5注释具有与人类基因相似的基因结构.
6、扩展数据图2:哺乳动物基因组进化
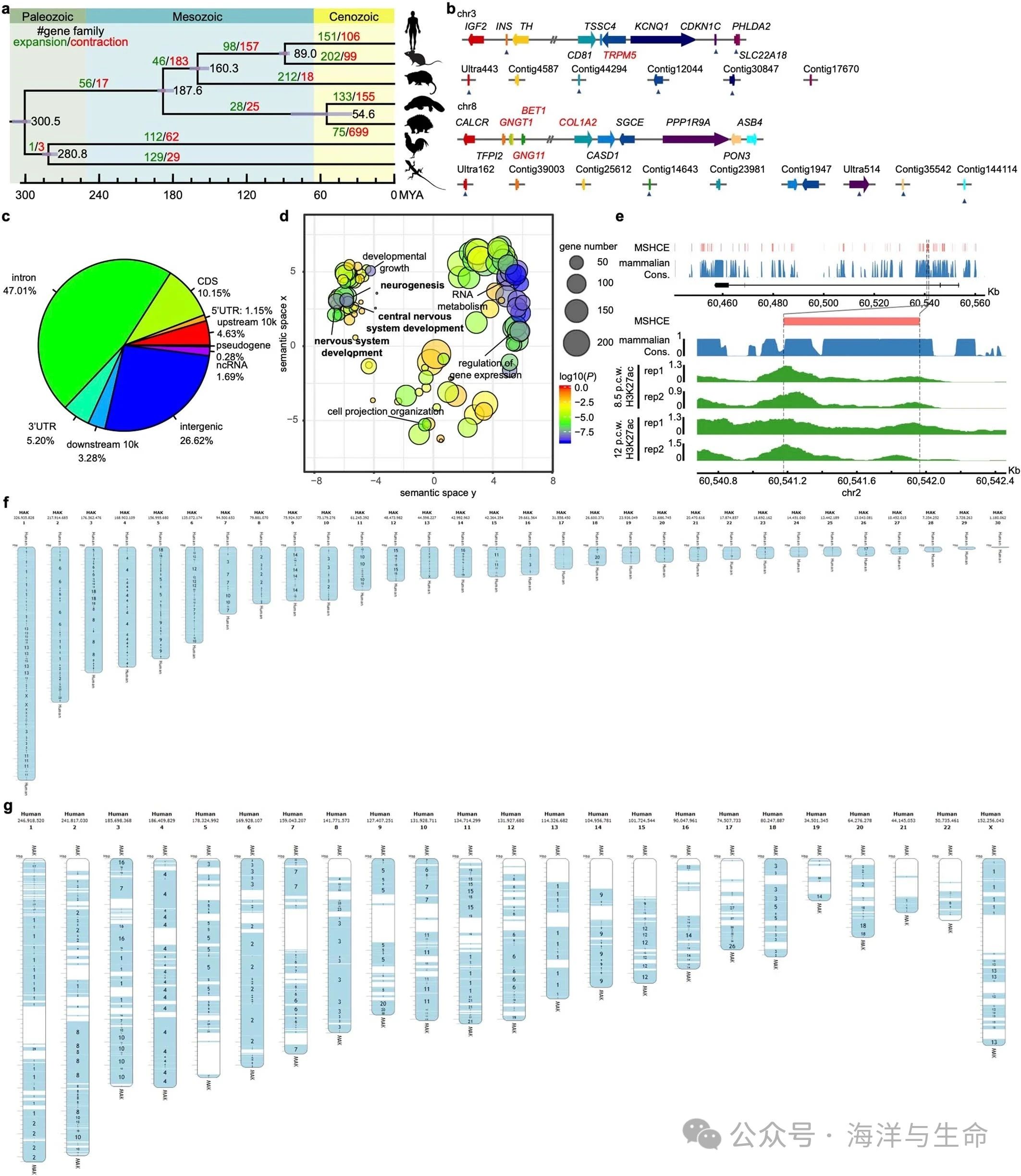
a,利用7个代表物种(人类、小鼠、负鼠、鸭嘴兽、针鼹、鸡和绿蜥蜴)中7946个一对一同源物的四重退化位点构建了系统发育树。用圆圈标记的节点的化石时间校准是从之前发表的一项研究67中获得的。在每个分支上标记了经历显著(Viterbi P<0.05)谱系特异性扩增(绿色)和收缩(红色)的基因家族的数量。
b,一些印迹基因簇的实例在mOrnAna1中与OANA5相比有所改善。每个同线性图的第一行表示mOrnAna1,第二行表示OANA5。已发现在人类和小鼠体内印记的基因的名称以黑色突出显示,非印记基因以红色突出显示。比对率低于70%的片段基因以三角形标记。双斜线表示长度超过100kb的中间区域。
c,MSHCEs在基因组元素上的分布。
d,在前300个MSHCE相关基因中富集GO术语。使用χ2检验计算富集的P值,并计算FDRs以调整多次检验。GO术语基于语义相似性进行聚类。与神经系统发育相关的GO术语以粗体突出显示。
e,在受孕后8.5周和12周,BCL11A中的一个MSHCE与从H3K27ac-ChIP-seq实验推断的增强子信号重叠(p..w.)。
f,以500kb分辨率重建的MAC的进化高速公路比较染色体浏览器可视化。覆盖在每个MAC上的块表示人类合成片段。区块内的数字表示同源的人类染色体。
g,进化路径以500kb分辨率对人类基因组进行比较染色体浏览器可视化,每个块覆盖在每个人类染色体上,代表祖先哺乳动物基因组的假定染色体片段。区块内的数字描绘了祖先哺乳动物的染色体数量。
7、扩展数据图3:单孔目动物免疫基因家族的进化

a,鸭嘴兽和针鼹的MHC基因位于两条不同的染色体上,但参与抗原呈递的经典I类和II类基因位于每个基因组的单个簇内。
b,代表性哺乳动物和鸡I类基因的系统发育关系。单孔目中的经典I类基因(红色)表现出高度相似性,这在其他物种中很少观察到。c,d,MHC II类α(c)和β(d)基因的系统发育关系。前缀为“HLA”、“Modo”、“Phci”、“Oran”、“Taac”和“Gaga”的基因分别表示人类、负鼠、考拉、鸭嘴兽、针鼹和鸡的基因。
e,鸭嘴兽(黄色)、针鼹(紫色)、考拉(绿色)、小鼠(橙色)、人类(红色)、绵羊(灰色)、奶牛(暗红色)和鸡(暗黄色)的推定功能Vγ序列之间的系统发育关系。根据之前的一项研究,90的类群显示在树的外部,假定的有袋动物-单孔目特定类群用“?”表示。
f,单孔目中β-防御素基因的协同保守性和针鼹中功能性毒液防御素的丧失。毒液防御素(OavDLP基因)和毒液样防御素(DEFB-VL基因)显示为红色。仅显示推定的功能性防御素。
g,针鼹中假定的OavDLP损失。在鸭嘴兽中,OavDLP基因和DEFB-VL各自包含两个外显子(用方框和三角形表示)。鸭嘴兽DEFB-VL的两个外显子都可以定位到针鼹染色体X2上。单个鸭嘴兽OavDLP外显子可以被定位到针鼹染色体X2上,而第二个外显子则不能。灰色链接表示鸭嘴兽-针鼹LASTZ排列。
h,DEFB-VL和OavDLP基因的系统发育关系表明,祖先的单孔目具有全部三个OavDLP基因,但针鼹已经失去了其中两个(OavDLP-B和OavDLP-C)。未显示分支长度。
8、扩展数据图4:单孔目性染色体的基因组组成
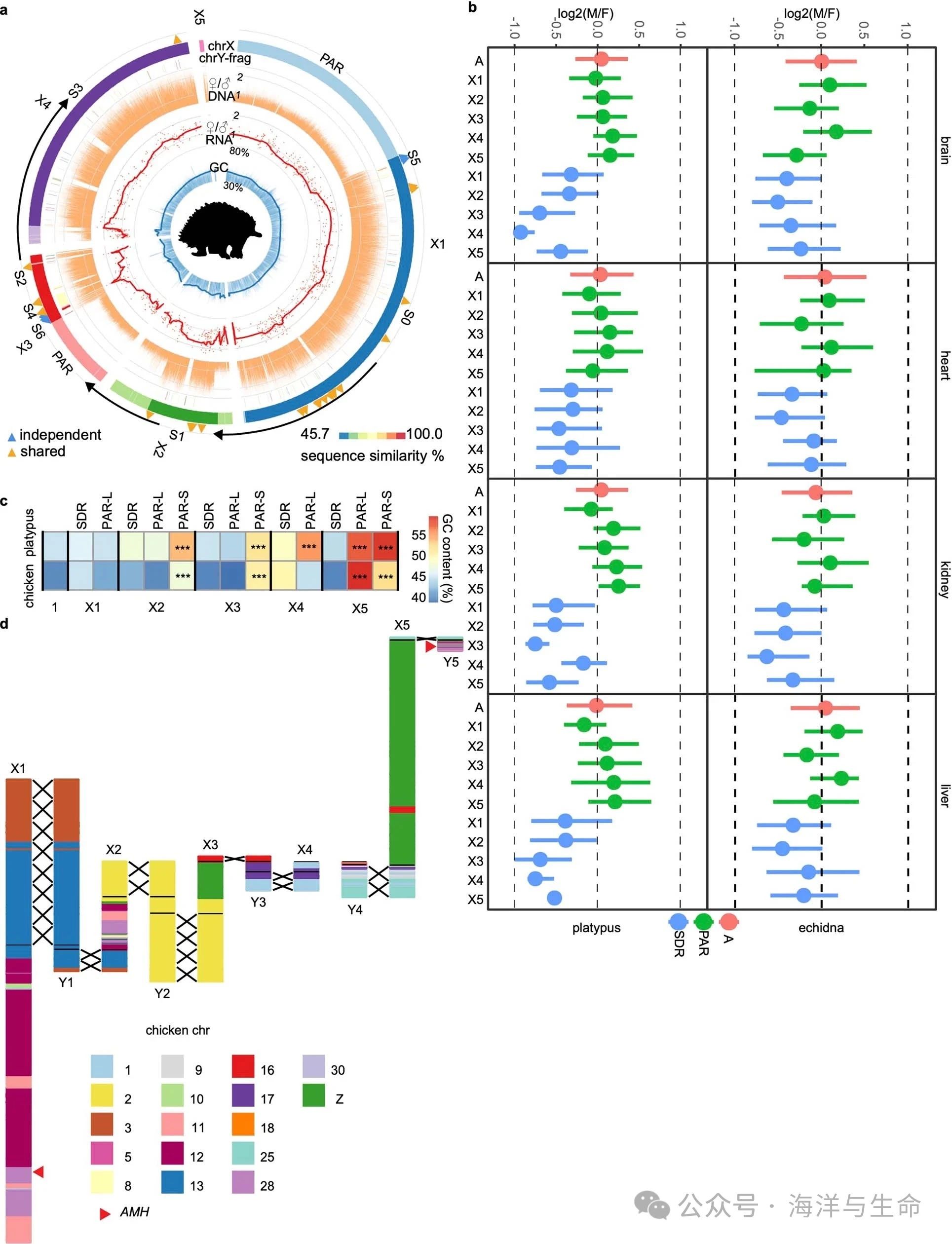
a,针叶树性染色体的组成。圆环图(从外圈到内圈)显示:X染色体的PAR显示为浅色,SDR显示为深色;组装的Y染色体片段显示与同源X染色体的颜色标度序列相似性水平;非重叠5kb窗口中Illumina DNA测序深度的归一化F/M比率;成年肾脏的F/M表达率(每个红点是一个基因)和平滑的表达率趋势;以及非重叠2kb窗口中的GC含量。此外,与X染色体具有相似序列差异的Y连接片段表明了进化阶层的模式。正如预期的那样,F/M DNA深度比在PAR处集中在1,但在SDR处约为2。一些PAR显示出比抑制X和Y之间重组的区域显著更高的GC含量。
b,单孔目的部分剂量补偿。四点范围图显示了鸭嘴兽和针鼹的大脑、肾脏、心脏和肝脏中雄性与雌性(M/F)表达比率的log2转换值。正如预期的那样,对于常染色体(A)和PAR上的基因,M/F表达比率的log2转化值接近0,而对于SDR上的基因来说,在所有组织中的表达都是女性偏向的,这表明单孔目具有部分剂量补偿。晶须表示第25–75个百分位数,圆圈表示中值。
c,有些PAR的GC含量明显高于SDR。对于鸭嘴兽,一些PAR(X2-PAR-S、X3-PAR-S-、X4-PAR-L、X5-PAR-S和X5-PAR-L(其中-S是染色体较短的标准杆数,-L是较长的标准杆数))的GC含量(1-kb非重叠窗口)显著高于同一染色体的SDR,SDR在热图中标记为星号。我们还检查了它们在鸡中的同源序列,作为染色体成为性染色体之前祖先地位的代表,并发现在鸡中PAR的同源区中GC含量同样高于SDR的同源区。
d,沿着每个鸭嘴兽性染色体的直源鸡片段图谱。鸭嘴兽X染色体和Y染色体之间的PAR由杂交表示。我们还标记了假定的性别决定基因AMH的位置。
9、扩展数据图5:鸭嘴兽和针鼹分化后PAR的进化
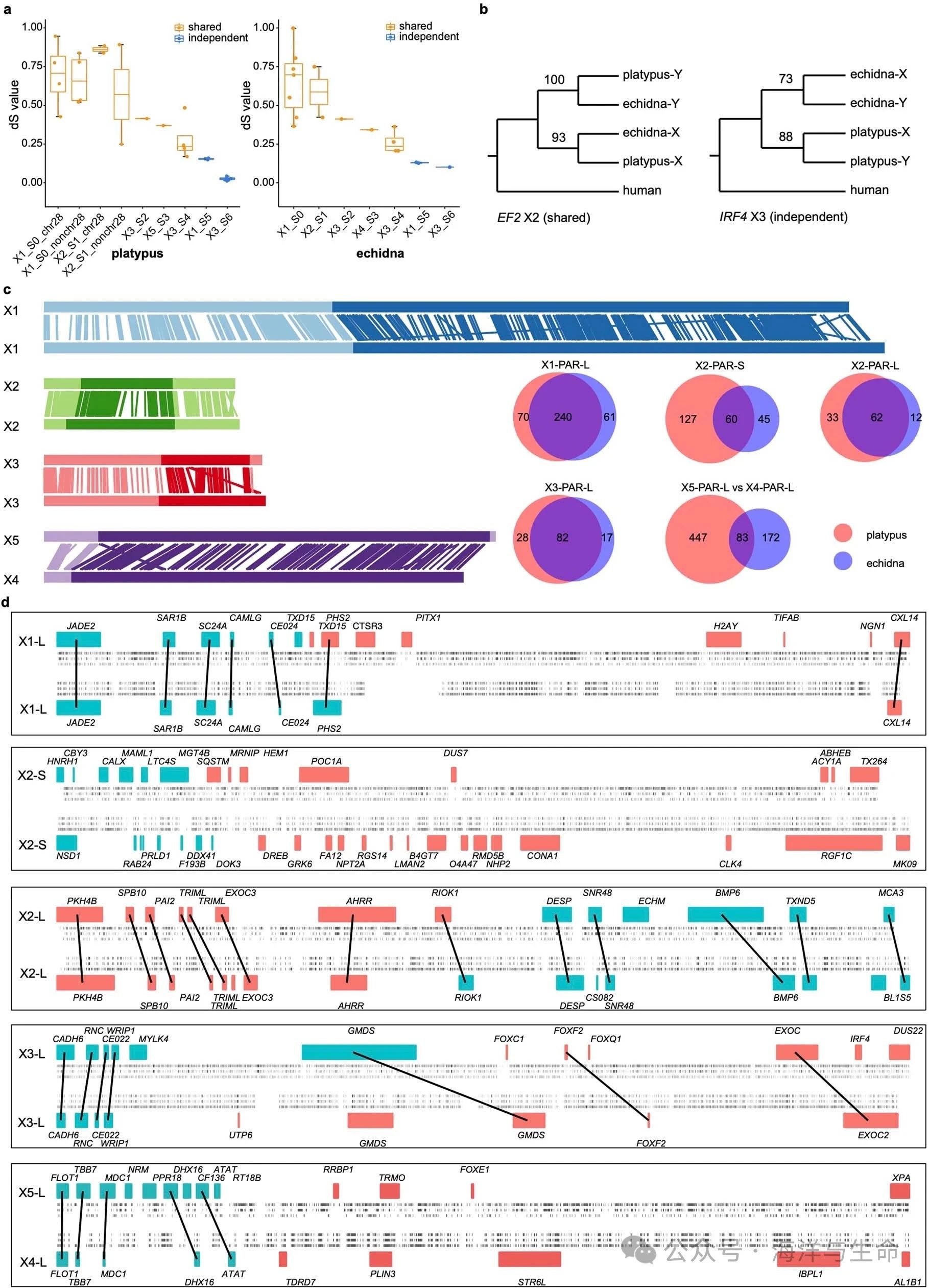
a,鸭嘴兽性染色体和针鼹性染色体成对dS值的分布。在鸭嘴兽和针叶树中,X1-S0区域的配子体对(图2)与鸡28号染色体基本同源,其dS值高于任何其他性别连锁区域的dS值。这表明X1-S0是最古老的进化阶层。因此,我们还显示了X2的鸭嘴兽基因与其他基因(X1_S0_chr28)在鸡28号染色体上具有同源物。根据不同染色体区域的dS值的顺序,我们推断出进化层形成的时间顺序,称为S0–S6。对于鸭嘴兽,从左到右绘制n=5、5、2、1、1、4、2和6个XY配子体对。对于针鼹,从左到右绘制n=7、2、1、1、4、2和1XY配子体对。方框图显示了中位数、四分位数(方框)和范围(胡须)。
b,单孔目的共同祖先(X2中的EF2)和两个单孔目物种(X3中的IRF4)中独立进化的配子体的系统发育树实例。
c,利用鸭嘴兽和针鼹X染色体的比对(PAR,浅色;SDR,深色;顶部染色体来自鸭嘴兽,底部染色体来自针鼹)来推断鸭嘴兽的X2-PAR-S和X5-PAR-L在分化后独立于针鼹进化而来,因为它们的长度不同。鸭嘴兽和针鼹之间的PAR基因文氏图支持这一点,其中大多数基因在独立进化的PAR中并不共享。
d,鸭嘴兽和针鼹之间PAR-SDR权边界的对齐。基因的排列(边界周围±1 Mb)支持鸭嘴兽和针鼹X2-PAR-S和X5-PAR-L的独立进化,因为它们的大多数基因在PAR–SDR边界(蓝色,PAR基因;红色,SDR基因;鸭嘴兽,顶部染色体,针鼹,底部染色体)上不同源。我们使用品系来连接这两个物种的基因,只要它们是直向同源的。对于每个X染色体,我们还标记了它们的重复信息。每个X对之间显示了六个重复轨迹,从上到下:鸭嘴兽的整体重复内容;鸭嘴兽的LINE/L2元件;鸭嘴兽的SINE/MIR元素;针鼹的SINE/MIR元素;针鼹的LINE/L2元素和针鼹的总重复含量。我们在PAR-SDR边界没有发现明显的重复富集,如之前在奶牛中所示。
10、扩展数据图6:单孔目的性染色体进化
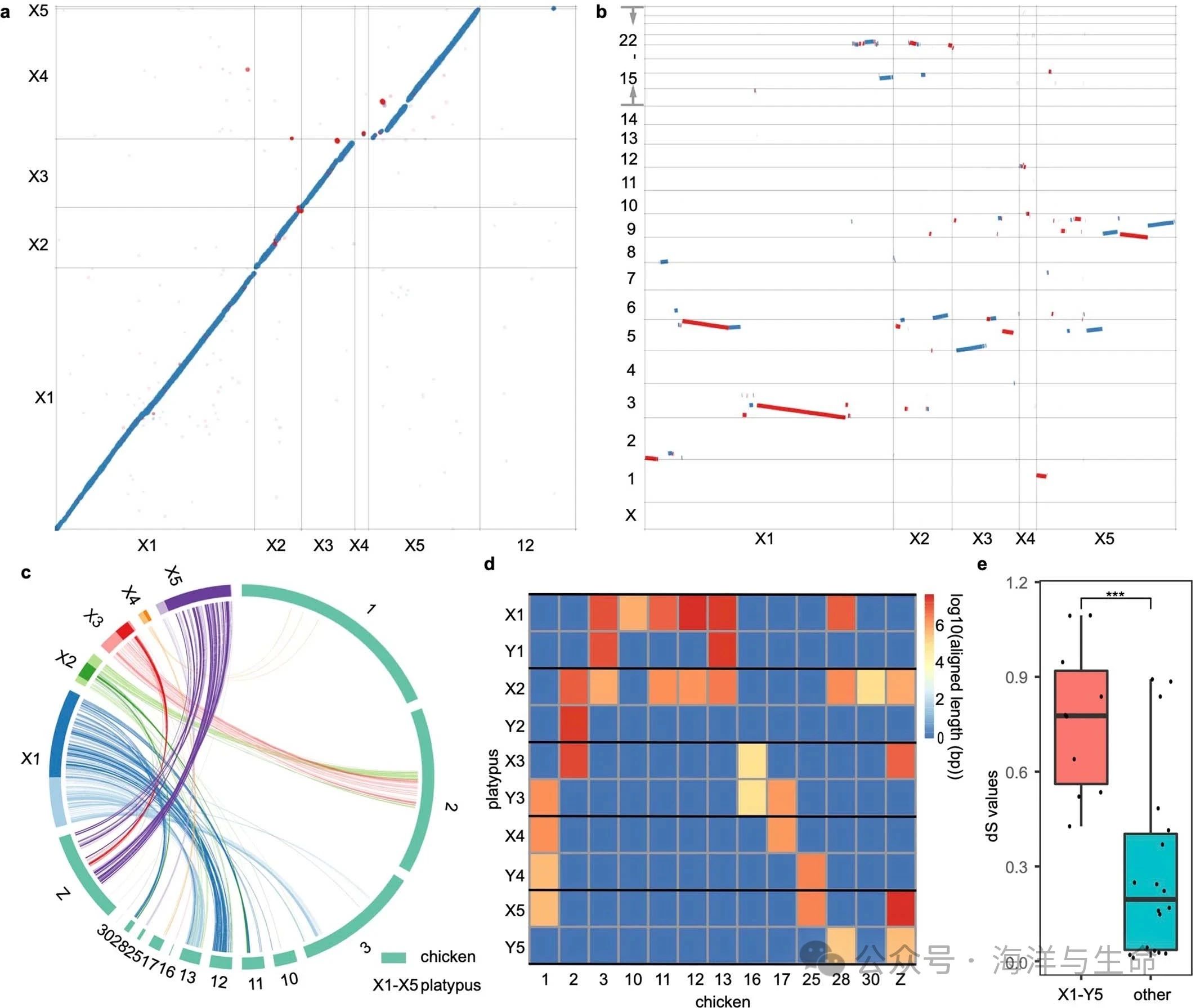
a,显示鸭嘴兽(x轴)和针鼹(y轴)x染色体同源性的Mummerplot。蓝色线条:向前对齐;红线:反向对齐。对于针叶树,X1、X2和X3分别与鸭嘴兽X1、X2、X3同源。棘皮动物X4与鸭嘴兽X5同源。对于针鼹X5,它与任何鸭嘴兽性染色体都不同源,而是与鸭嘴兽12号染色体同源。
b,鸭嘴兽X染色体(X轴)和人类染色体(y轴)之间的同源性
c,鸭嘴兽性染色体与鸡的同源关系
d,鸭嘴兽和鸡之间的排列显示了鸭嘴兽性染色体链的交替配对模式。
e,X1–Y5对(n=18)和其他性染色体对(n=10)配子体之间的X/Y成对dS比较。方框图显示了中位数、四分位数(方框)和范围(须)。
11、扩展数据图7:单孔目性染色体的染色质构象。

a,鸭嘴兽性染色体之间的Hi-C相互作用,其中1号染色体显示为对照。
a,b,Hi-C数据(a)检测到鸭嘴兽性染色体之间存在意外的染色体间相互作用(以红色显示),而其他染色体(b)的大多数相互作用都在相同的染色体内(以红色表示)。
c,鸭嘴兽性染色体之间的Hi-C染色体间相互作用(inter_XY,n=2711 100kb窗口)显著高于常染色体之间的Hi-C染色体间交互作用(inter_A,n=14342930 100kb窗口)。方框图显示了中位数、四分位数(方框)和范围(须)
d,Y2和Y3之间的相互作用强度高于Y2和其他染色体之间的相互作用力强度。n=1002、228、5025、67313和6904867 100kb窗口分别显示在Y2-Y2、Y2-Y3、Y2-其他例如chr、Y2-A和A-A中。
e,推测鸭嘴兽性染色体系统的三维结构。X染色体显示为红色,Y染色体显示为蓝色,PAR显示为浅色。由Hi-C推断的染色体间相互作用用虚线表示。
f,Hi-C相互作用揭示了针鼹性染色体之间出乎意料的染色体间相互作用。
g,假定的CTCF结合位点在鸭嘴兽和针鼹性染色体的TAD边界富集。对于鸭嘴兽的每个X染色体,我们计算了它们每10kb的假定CTCF结合位点密度,并沿着±500kb的TAD边界绘制了它们的图。由于检测到少于10个TAD边界,因此没有显示鸭嘴兽X4和针鼹X5。
h,推定的CTCF结合位点密度图显示其在鸭嘴兽、针鼹、人和鸡的同源区域中富集。
12、扩展数据图8:单孔目中饮食相关基因的缺失
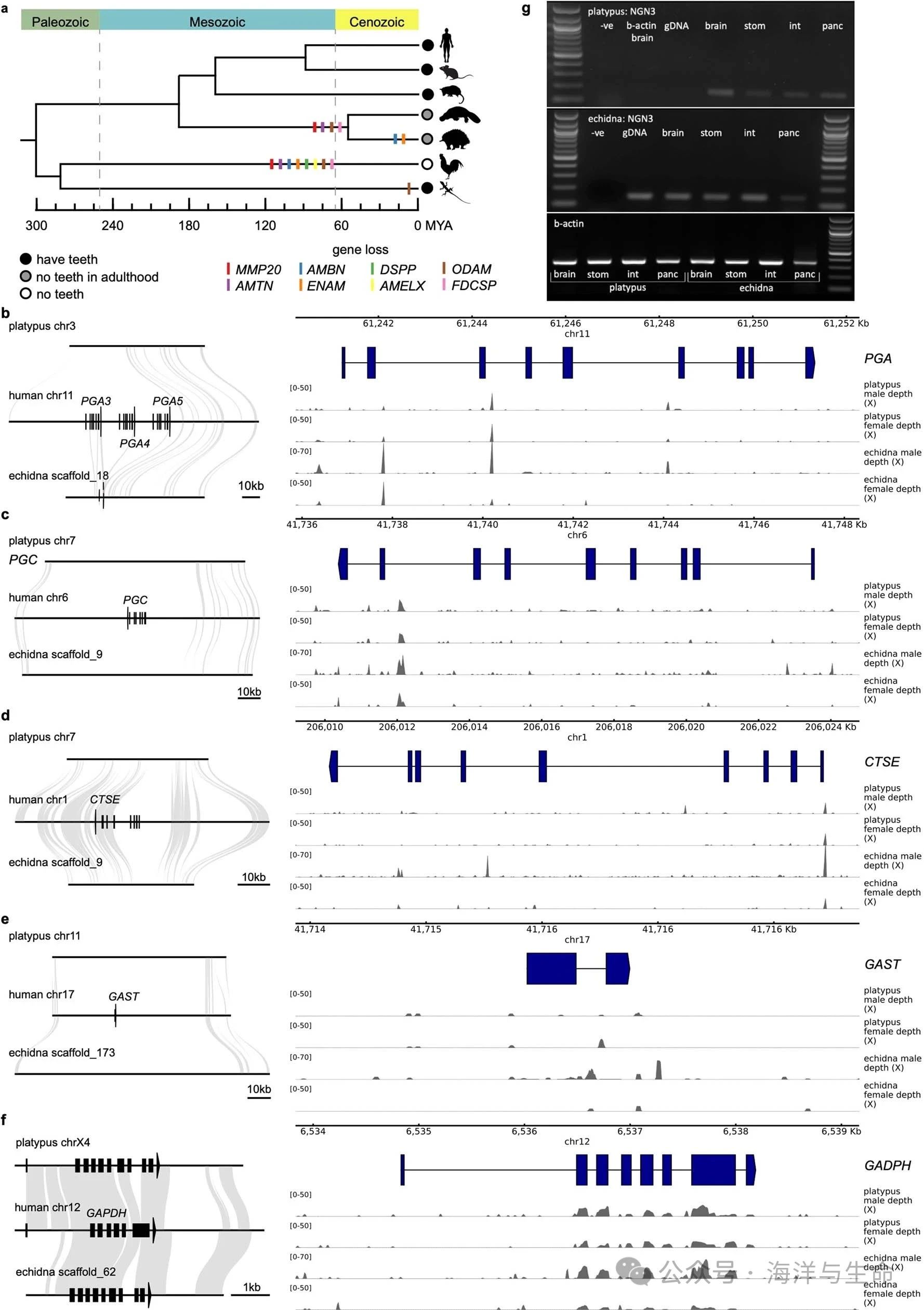
a,代表性哺乳动物和爬行动物的牙齿相关基因缺失。
b-f,通过全基因组比对和读取图谱显示,两种单孔目中消化相关基因的潜在损失。在每个面板中,共线性图中有三条线,从上到下分别代表鸭嘴兽、人类和针鼹基因的同源区。灰色链接表示人类-鸭嘴兽和人类-针鼹LASTZ比对。每个矩形或三角形代表一个外显子。基因片段用虚线表示。鸭嘴兽和针鼹的Illumina读数与鸭嘴兽或人类基因组对齐(Ensembl release 87),每个基因的侧翼区域通过pyGenomicTrack可视化。GAPDH区域也被绘制为对照。
g,RT–PCR表达分析显示NGN3在鸭嘴兽和针叶树的脑、胃、肠和胰腺中的表达。这些结果与其他哺乳动物相似。这与测序结果一起表明,单孔目中存在NGN3,并且可能功能正常。NGN3、NGN3引物;β-肌动蛋白引物;-ve,阴性对照,无模板;gDNA,基因组DNA模板;脑、脑cDNA模板;胃cDNA模板;int,肠cDNA模板;panc、胰腺cDNA模板。泳道1(顶部)、1、8(中间)和9(底部)是100bp的DNA梯状条带:1517、1200、1000、900、800、700、600、500/517、400、300、200和100bp。鸭嘴兽中NGN3的PCR产物的预期大小为157bp,针鼹的PCR产物为145bp,β-肌动蛋白基因组区的PCR产物是597bp,cDNA是348bp。
13、扩展数据图9:单孔目动物的味觉受体进化和嗅觉受体组织
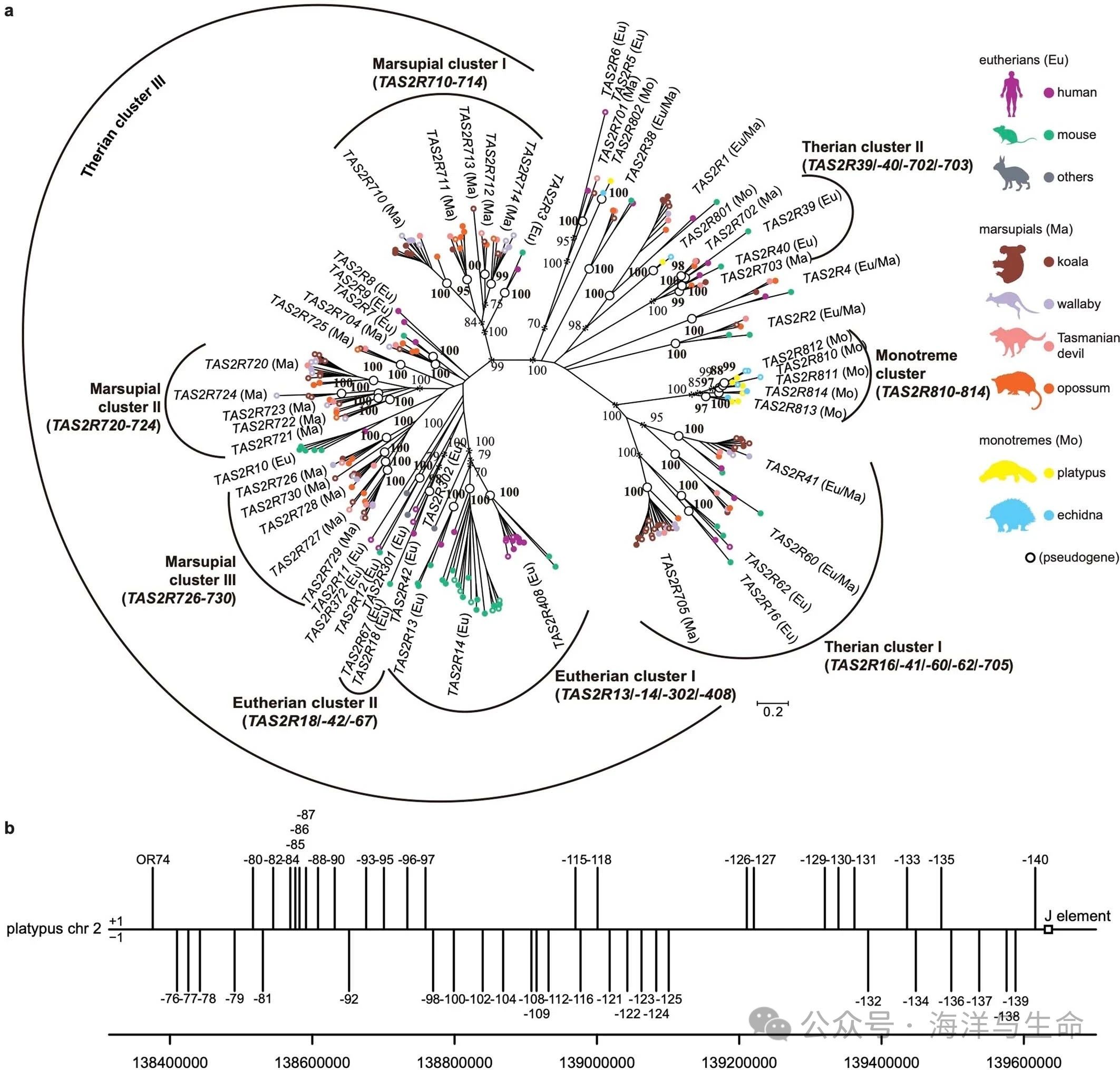
a,苦味受体的最大可能性哺乳动物广泛基因树(TAS2R基因)。共有28个真骨类(Eu)、27个有袋类(Ma)和7个单孔目特异性(Mo)直系同源基因组(由≥95%的bootstrap值支持),其中直系同源基因群分支的节点用白色开放圆圈表示。在连接同源基因组分支的节点中,Bootstrap值≥70%用星号表示。有3个兽属(I、II和III),2个真兽属(Ⅰ和II),3个有袋动物(I、Ⅱ和III)和一个单孔目特有的集群,在这些集群中,每个分类单元的共同祖先在与其先前祖先分离后发生了大规模扩张事件。
b,鸭嘴兽2号染色体(138375798–139616970 bp)上跨越1.2 Mb的完整I类嗅觉受体(OR)簇的基因组组织。垂直线表示48个完整的I类OR基因。白色开口框表示J元件,这是哺乳动物I类OR簇(染色体2:139639465–139639907 bp)的一种推测顺式调节元件(增强子)。
14、扩展数据图10:单孔目中与血红蛋白清除和繁殖相关的基因组特征
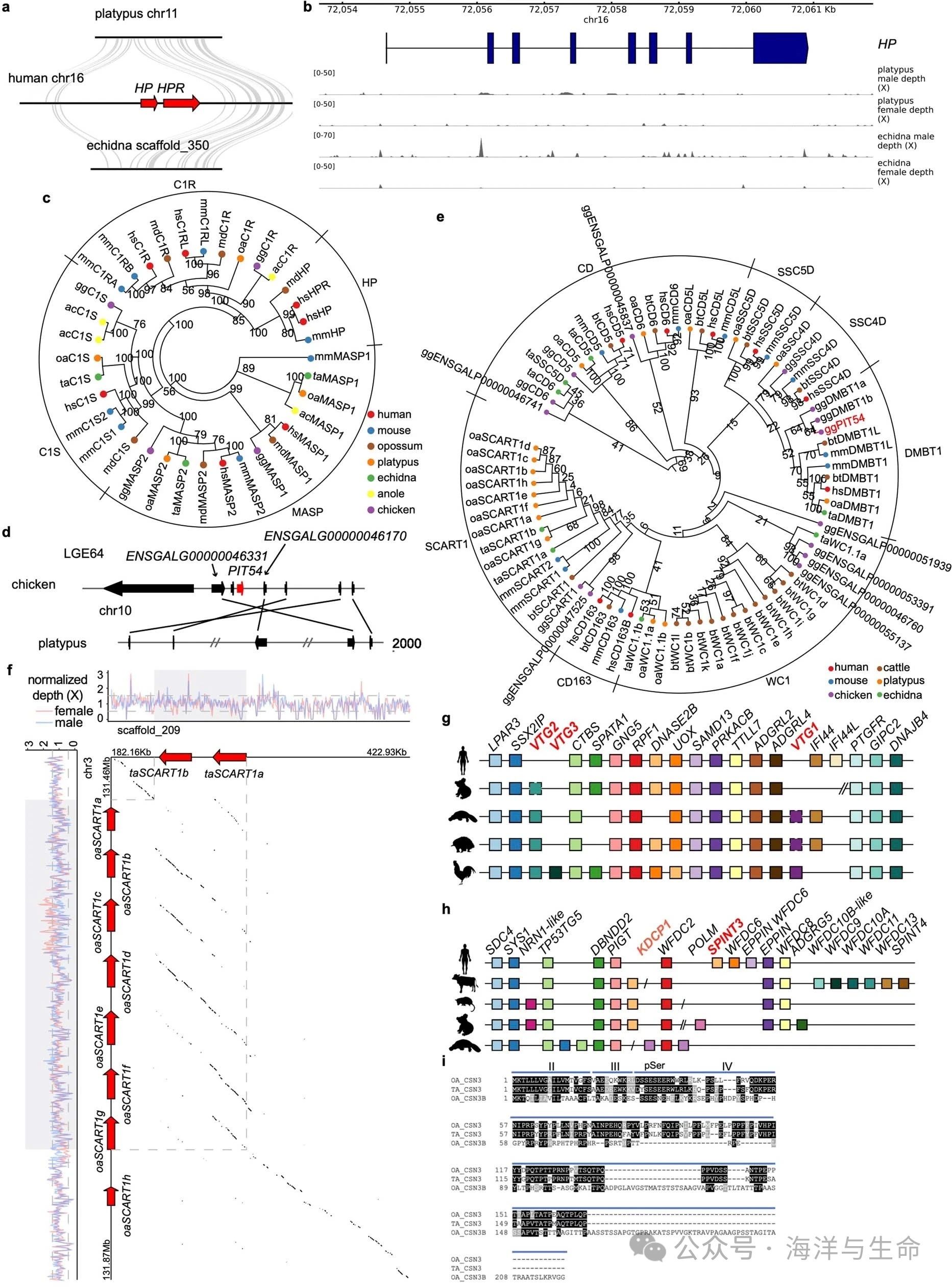
a,b,通过全基因组比对(a)和读取图谱(b)确认单孔目中HP的缺失。灰色线条表示人类-鸭嘴兽和人类-针鼹LASTZ比对。鸭嘴兽和针鼹的Illumina读数与人类基因组对齐(Ensembl release 87),HP的编码区通过pyGenomicTrack可视化。HP外显子的覆盖范围有限,这表明单孔目中没有HP
c,使用最大似然法研究不同物种HP和相关蛋白酶的系统发育树。
d,鸡和鸭嘴兽之间PIT54区域的基因同源性图。图中没有显示棘皮动物,因为PIT54的侧翼直系同源物位于不同的支架上,使我们无法通过同源性来确定该基因的存在。
e,使用相邻连接方法的B族清除剂受体富含半胱氨酸家族成员在不同物种之间的系统发育树。
f,通过鸭嘴兽和针鼹之间SCART1同源区的点图和作图深度来确认SCART1数量差异。鸭嘴兽的SCART1簇的区域沿x轴绘制,而针鼹的序列沿y轴绘制。根据两个物种之间的LASTZ排列,点图中的线被可视化。沿着每个序列的标准化雄性和雌性读取深度在500bp窗口中计算,并沿着每个序列绘制。两性的标准化深度,特别是在阴影区的深度,沿着两个物种都集中在1,这证实了两个物种之间的SCART1数量差异是真实的,而不是由于组装问题。
g,卵黄蛋白原基因的协同保守性。卵黄蛋白原(VTG)基因VTG1、VTG2和VTG3周围区域的协同保守性。假基因用虚线标出。单孔动物具有假基因VTG1、功能性VTG2和无VTG3;考拉中存在一个假基因VTG2。显示了人类(H. sapiens)、考拉(Phascolarctos cinereus)、鸡(Gallus Gallus)、鸭嘴兽(O.anatinus)和针鼹(T.acuretus)的共生图。
h,协同保守区域包含SPINT3。Kunitz型丝氨酸肽酶抑制剂3(SPINT3)周围区域的协同保守性。在鸭嘴兽中没有检测到SPINT3的拷贝,但该区域的许多其他侧翼基因是保守的。检测到具有WFDC结构域的其他成员,包括未与任何已知基因(标记为KDCP1)对准的两个Kunitz结构域成员。据报道,人类(H. sapiens)、奶牛(B. taurus)、灰色短尾负鼠(Monodelphis domestica)、考拉(Phascolarctos cinereus)和鸭嘴兽(O.anatinus)的共生图。
i,单孔目动物酪蛋白3(CSN3)蛋白序列比对。在单孔目中鉴定的所有三种CSN3蛋白都具有CSN3的经典五个外显子结构,具有未翻译的外显子I和IV(未显示)、外显子II中的信号肽、编码11个残基的小外显子III、在外显子IV的5′端的pSER簇(S**)和相对大的富含P/Q的外显体IV。OA, O. anatinus(鸭嘴兽);TA, T. aculeatus(短吻针鼹)
【文章链接】https://www.nature.com/articles/s41586-020-03039-0
文章来源:海洋与生命公众号



 京公网安备11010102006642号
京公网安备11010102006642号