Nature 重磅!EEE:猿类基因组的完整测序
发布时间:2025年04月18日
科学家们已成功测序六种现存猿类物种的完整基因组,使得此前难以组装的基因组区域得以进行期待已久的比较。猿类,包括人类,是我们在进化上最亲近的亲属。比较人类与其他猿类的基因组对于理解人类基因组的功能和我们的进化历史至关重要。然而,由于猿类基因组庞大且包含重复序列,许多基因组区域一直难以准确测序和重建,导致迄今为止的比较受限。在《自然》杂志上,Yoo等人报告了六种猿类物种的完整基因组序列,代表了所有主要的猿类谱系:黑猩猩(Pan troglodytes)、倭黑猩猩(Pan paniscus)、大猩猩(Gorilla gorilla)、婆罗洲猩猩(Pongo pygmaeus)、苏门答腊猩猩(Pongo abelii)和合趾猿(Symphalangus syndactylus)。

在2003年首个人类基因组序列完成后不久,黑猩猩的基因组组装也被发布。随后,其他大型猿类的基因组组装也相继完成,如大猩猩、苏门答腊猩猩和倭黑猩猩,以及与人类关系较远的小型猿类。这些基因组为编目猿类进化过程中积累的遗传差异提供了宝贵的机会,包括人类特有的变化。然而,由于这些最初发布的基因组是不完整的草图,比较只能在正确解析的基因组部分进行。因此,这些研究主要集中在相对较小的差异上,排除了极度重复的序列和大规模的结构差异,如基因组序列的倒位和重复。

Fig. 1: Chromosomal-level assembly of complete genomes for great apes.
Yoo等人的工作将猿类基因组序列的质量提升到与现有人类序列相同的水平,使我们能够研究几乎整个人类基因组的进化历史。这一宝贵资源还有助于对先前无法访问的基因组区域进行比较分析,其中许多具有生物医学相关性。

Fig. 2: Divergent regions and repeats.
新鉴定的区域包括指导细胞分裂的染色体部分。这些区域称为着丝粒,将每条染色体分为两臂。着丝粒由称为α-卫星的小型重复序列组成,α-卫星重复的模式本身可以形成所谓的高阶阵列。尽管单个着丝粒可以达到数百万个核苷酸碱基,但高阶阵列的序列组织尚不完全清楚。
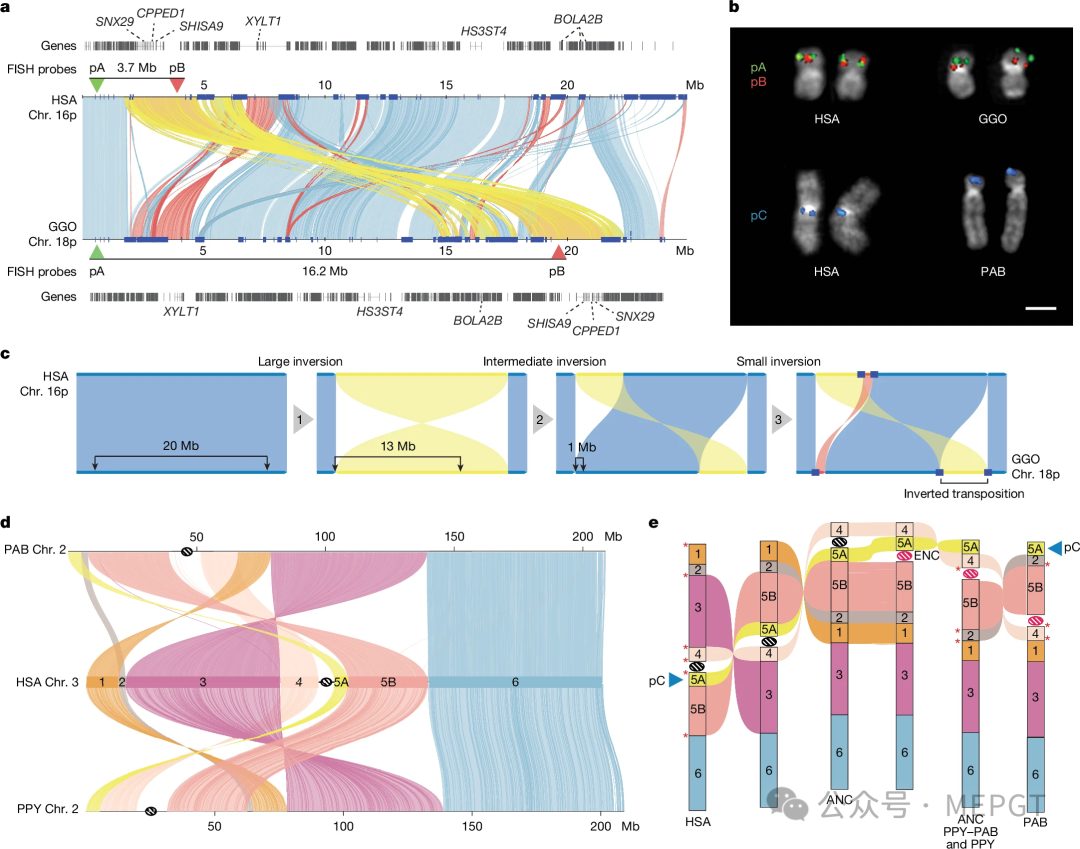
Fig. 3: Inversions and evolutionary rearrangements in great apes.
在他们的研究中,Yoo等人描述了大多数猿类染色体的完整着丝粒组成。他们描述了物种之间和物种内部在长度和序列组成上的广泛变异,这部分是快速和近期进化的结果。例如,倭黑猩猩中约40%的着丝粒在最多一百万年前减少了约300倍,导致该谱系特有的“迷你着丝粒”。类似地,尽管婆罗洲和苏门答腊猩猩在相对较近的96万年前才分化,但约五分之一的婆罗洲猩猩染色体包含新出现的高阶阵列,而苏门答腊猩猩则没有,体现了这些区域在短暂进化时期内的变化。
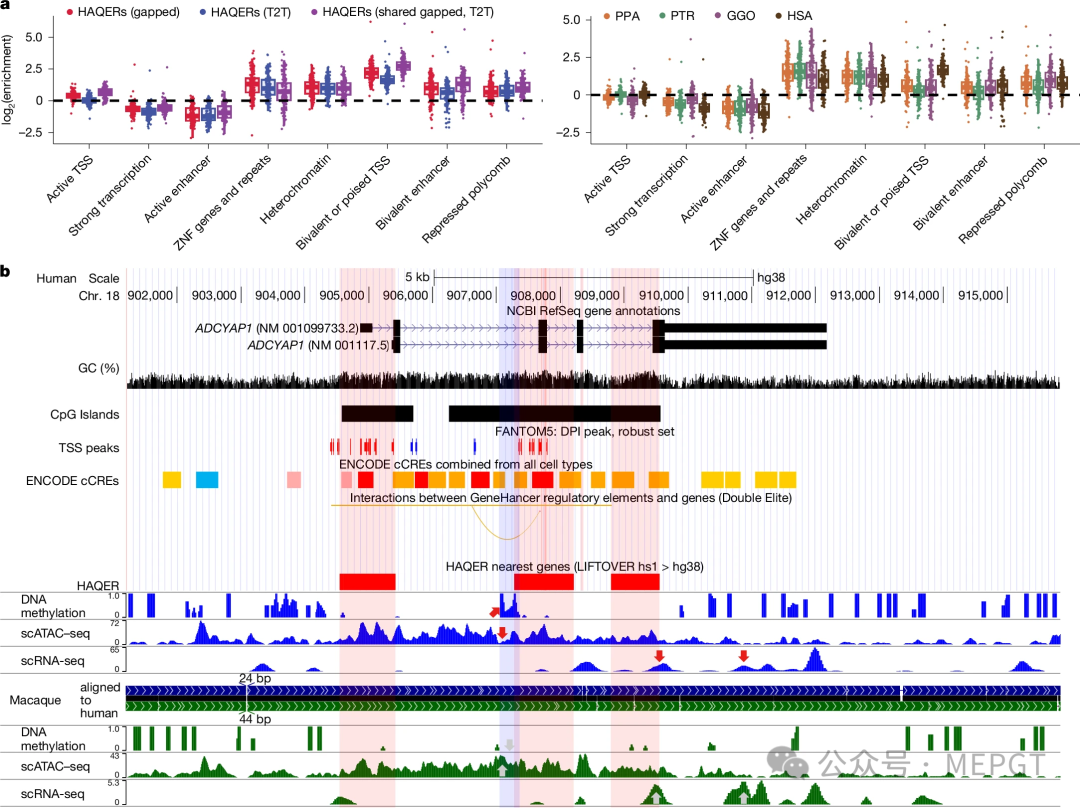
Fig. 4: AQERs.
一些染色体(在人类中为13、14、15、21和22号)是近端着丝粒染色体,这意味着着丝粒靠近染色体的一端。在人类中,近端着丝粒染色体的短臂几乎不携带基因,除了编码构建核糖体所需的核糖体RNA的基因。核糖体RNA基因的多个拷贝以大的阵列形式存在于核仁组织区域(NORs)中,周围环绕着重复序列。由于其重要性,核糖体RNA基因在进化上是保守的,但Yoo等人显示它们在拷贝数上高度可变,并描述了周围区域的广泛结构变异,通常导致序列的完全变化。

Fig. 5: Organization and sequence composition of the ape acrocentric chromosomes.
作者还观察到,不同物种的NORs数量存在差异。例如,合趾猿只有一个,而猩猩有十个。在猿类进化过程中,NORs的位置发生了变化,这意味着它们所在的染色体在不同物种中有所不同。这可能是由于异源重组(两个染色体或区域之间不均等的DNA交换)过程,这有助于通过消除重复区域拷贝之间的差异来维持NORs的序列和功能。其结果是所谓的协同进化现象,即不同拷贝的基因以相似的方式进化。作者显示,在同一物种内,协同进化可能发生在不同的NORs之间,也发生在同一阵列中核糖体RNA基因的拷贝之间,这些拷贝彼此之间比与其他NORs中的对应基因更相似。这些观察体现了研究所产出宝贵的资源价值,提供了描述和理解许多极其多样和不同基因组区域进化的工具包。
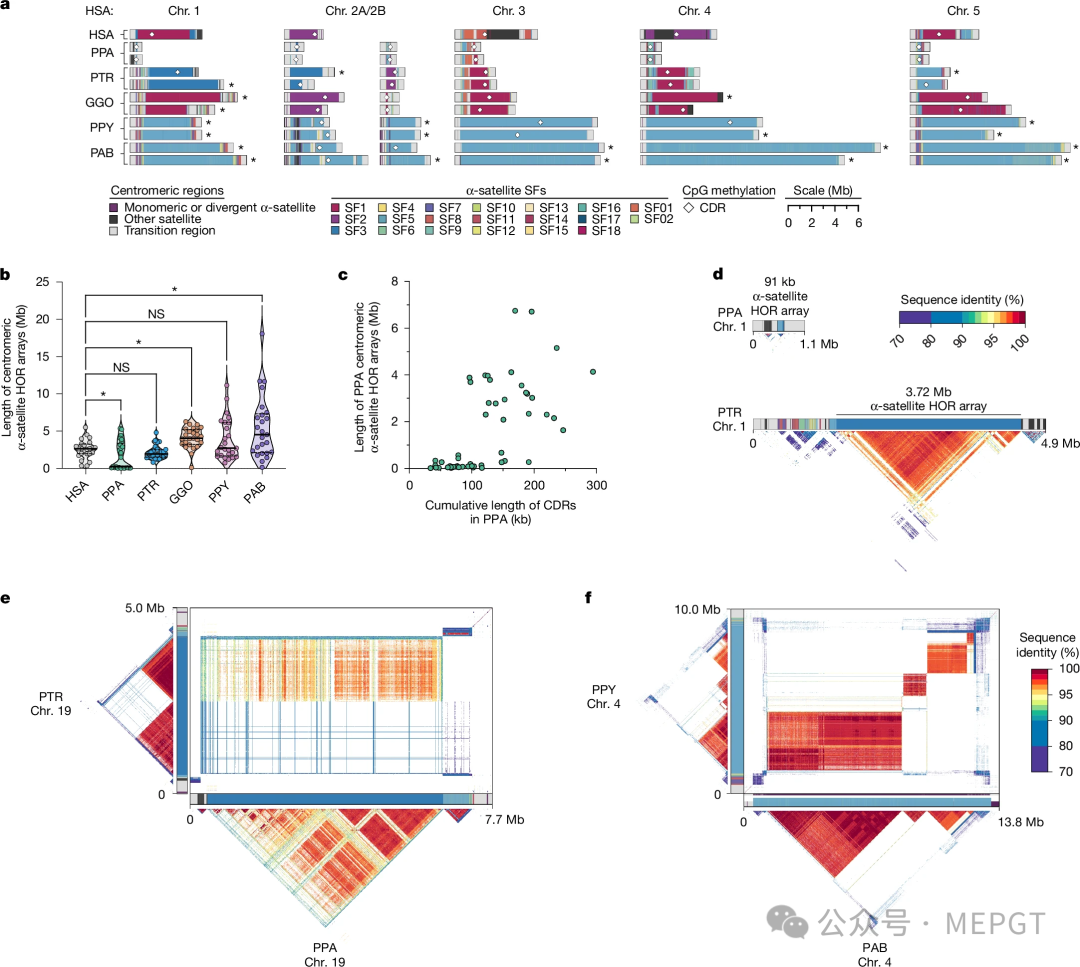
Fig. 6: Assembly of 237 NHP centromeres reveals variation in α-satellite HOR array size, structure and composition.
Yoo等人还分析了片段重复。这些基因丰富的重复基因组区域可以长达数千到数百万个碱基,包含具有高序列相似性的基因拷贝。片段重复很重要,因为它们是多种人类疾病的基础,并且在塑造大型猿类的进化中起到了关键作用。例如,人类特有的某些基因拷贝数增加被认为与额叶皮层体积的扩张有关。

传统上,大型猿类基因组中最具动态性和重复性的区域常被排除在比较研究之外,导致我们对人类进化的理解并不完整。在本研究中,作者提供了六种猿类物种的单倍型分辨率的参考基因组和比较分析结果:黑猩猩、倭黑猩猩、大猩猩、婆罗洲猩猩、苏门答腊猩猩和合趾猿。研究实现了染色体水平的高质量组装,序列精度极高(每270万个碱基不到1个错误),并完整测定了215条无缺口染色体,实现从端粒到端粒的覆盖。
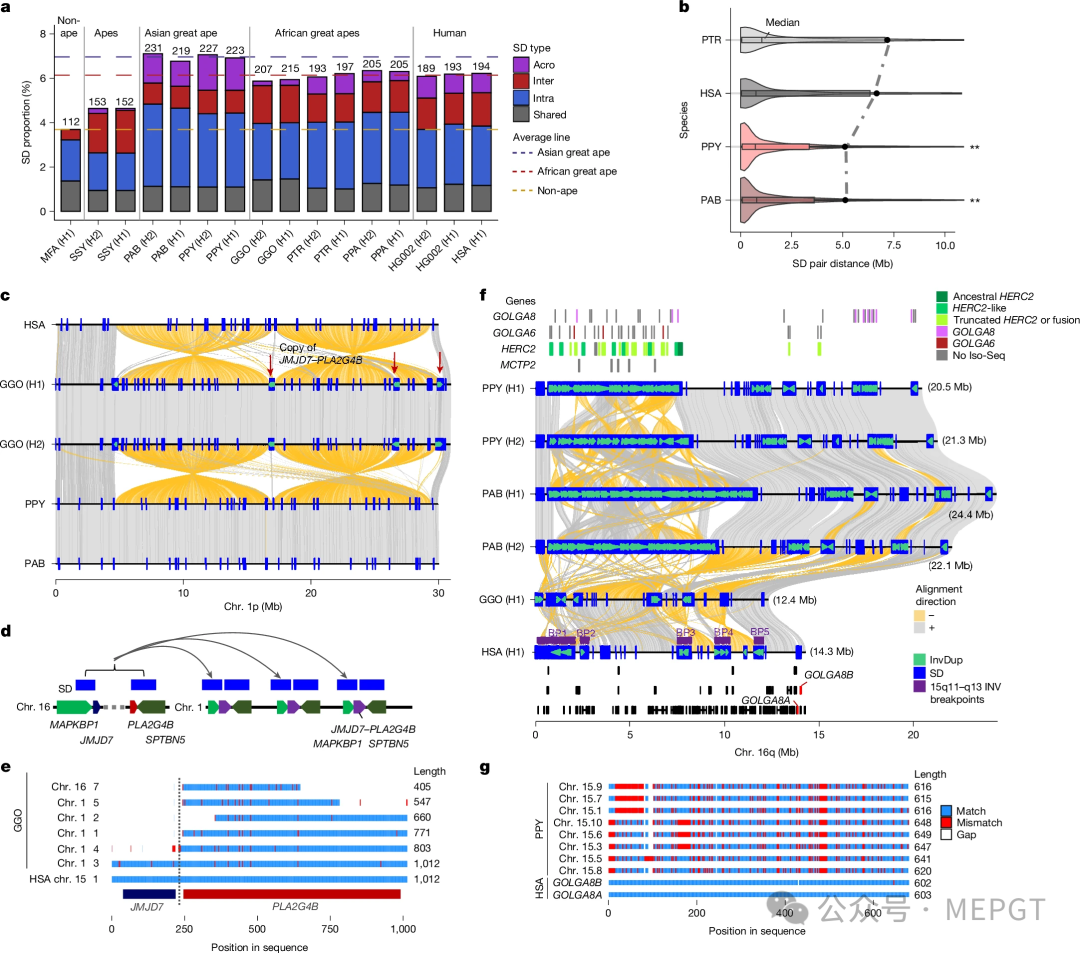
研究还解析了诸如主要组织相容性复合体(MHC)和免疫球蛋白基因位点等以往难以组装的区域,从而提供了深入的进化见解。比较分析使我们能够在不依赖人类参考基因组比对的前提下,研究此前未被表征或研究不完全的区域的进化与多样性。这些区域包括谱系特异的片段重复中新形成的基因家族、着丝粒DNA、近端着丝粒染色体以及亚末端异染色质等。该研究成果为未来人类与我们最亲近的现存猿类亲属的进化研究提供了全面且可靠的基础资源。

文章来源:公众号MEGBT